编辑 | 白菜叶
机器学习在精确快速预测结合亲和力方面展现了巨大的潜力。然而,现有模型的稳健性评估不足,未能有效完成在先导化合物优化过程中所需解决的任务,例如对一系列同类配体的结合亲和力进行排序,从而限制了其在药物发现中的应用。
牛津大学的研究团队首次提出了一种新的基于注意力机制的图神经网络模型,命名为AEV-PLIG(原子环境向量-蛋白质-配体相互作用图),以应对上述挑战。此外,他们还构建了一种更新颖且更贴近实际的分布外测试集(out-of-distribution test set),称为OOD测试。
研究人员在包含CASF-2016数据和用于自由能扰动(FEP)计算的测试集上对该模型进行了基准测试,显示出AEV-PLIG的竞争性能,并为以物理为基础的机器学习模型提供了实际的评估框架。
团队还展示了如何利用增强数据(如通过基于模板的建模或分子对接生成)显著提升结合亲和力预测的相关性以及在FEP基准测试中的表现排名。
该研究以《Narrowing the gap between machine learning scoring functions and free energy perturbation using augmented data》为题,于2025年2月8日发表在《Communications Chemistry》期刊上。

预测蛋白质与配体结合时自由能的变化是计算小分子药物发现的基础。这一过程在命中识别阶段至关重要,旨在识别对目标具有高亲和力的结合物,同时在从命中到先导化合物和先导优化过程中,需要对结合亲和力及与安全性和生物活性相关的诸多特性进行优化。
尽管机器学习算法和架构可以预测这些特性,但因缺乏相关的数据,其应用受到限制。基于结构的方法需依赖准确的结合亲和力测量和高分辨率解析的蛋白质-配体复合物(尤其是结合口袋)的三维(3D)结构。
增加训练数据的数量和多样性的一种策略是采用数据增强,生成合成数据以模拟真实世界的观察,或通过修改现有实验所得到的示例。这类方法在计算机视觉和自然语言处理等领域已被证明是有效的。
然而,由于某些固有的复杂性和物理化学的限制,从头生成有意义的生物和化学数据可能面临挑战。
新策略
在最新研究中,牛津大学的研究人员提出多种策略以增强机器学习(ML)评分功能的适用性,并对其性能提供更切实的评估,尤其关注药物发现领域的应用。
他们提出了一种新颖的结合亲和力预测方法AEV-PLIG,该方法结合了原子环境向量(AEV)和蛋白质-配体相互作用图(PLIG),采用注意力GNN架构以捕捉决定结合亲和力的复杂相互作用。
 图示:AEV-PLIG架构。(来源:论文)
图示:AEV-PLIG架构。(来源:论文)
研究人员对AEV-PLIG与RFScore、Pafnucy、OnionNet-2、PointVS、SIGN和AEScore进行了评估,采用多种针对OOD数据(OOD测试)和药物相关系统(FEP基准)或造成记忆惩罚(0配体偏差)的性能基准。结果显示,AEV-PLIG的性能明显落后于CASF-2016等广泛使用的评分函数基准。
在对不同的机器学习模型进行比较时,AEV-PLIG的表现良好,但没有任何模型在所有基准测试中均取得显著更优的性能,这突显了对多样化测试用例的需求,以有效评估新模型的特性与架构。
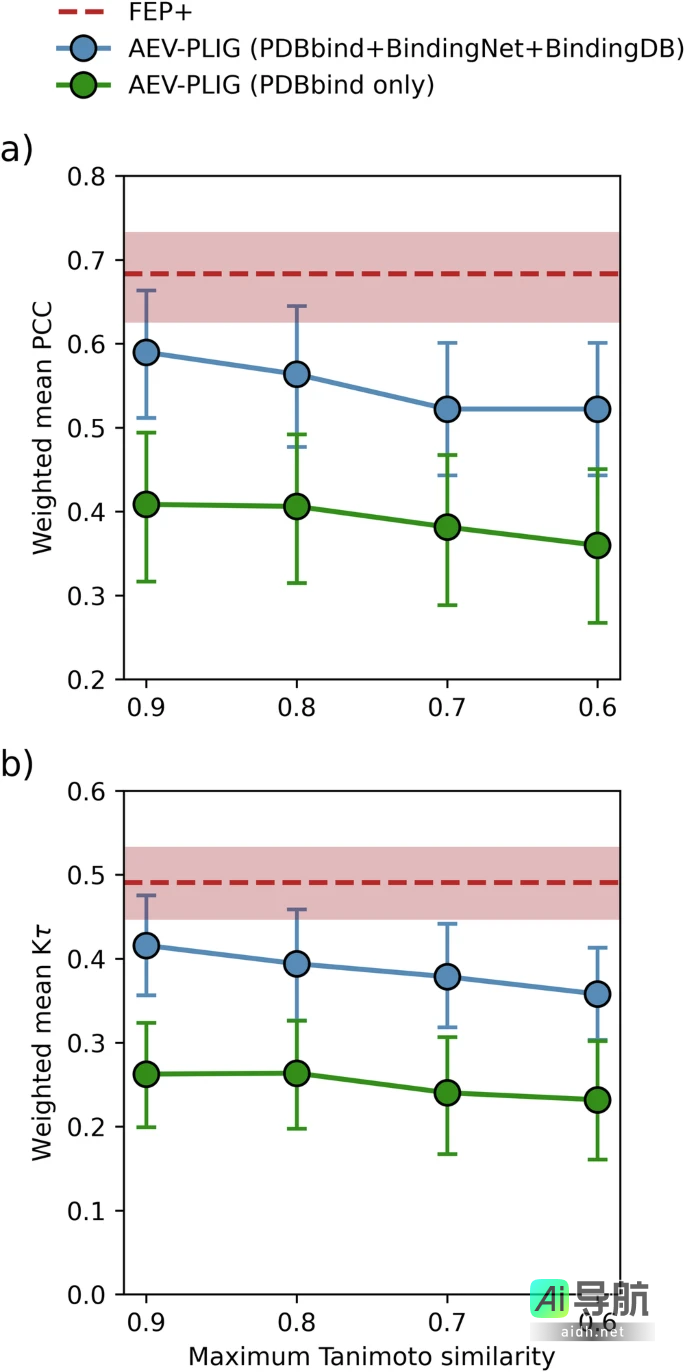
图示:AEV-PLIG模型在FEP基准测试中的表现。(来源:论文)
这些策略共同缩小了与FEP计算性能的差距(FEP+在FEP基准上的加权平均PCC和Kendall的τ分别达到了0.68和0.49),同时速度提高了约400,000倍。
这些测试集最初设计用于对FEP性能进行基准测试和验证,它们不仅能够为计算辅助药物设计(CADD)应用提供更真实的预测准确性评估,也为当前基于机器学习和分子动力学的方法提供了直接的比较依据。
 图示:FEP+与AEV-PLIG模型在FEP基准中针对具有25个或更多配体的目标进行性能比较。e5c1da0″ data-type=”tech_tasks”>药物发现系统中的排名能力。这是通过引入微小的结构变化来设计化合物,以增进其对特定生物靶点的结合亲和力。
图示:FEP+与AEV-PLIG模型在FEP基准中针对具有25个或更多配体的目标进行性能比较。e5c1da0″ data-type=”tech_tasks”>药物发现系统中的排名能力。这是通过引入微小的结构变化来设计化合物,以增进其对特定生物靶点的结合亲和力。
研究人员指出,额外的增强数据能够进一步提升未来的性能。随着诸如 AlphaFold 3、Umol 及 NeuralPlexer 等准确的(蛋白质-配体)结构预测模型的引入,这些数据未来将迅速变得更加可用。
结语
综上所述,AEV-PLIG 在多项基准测试中表现出与众多其他基于机器学习的方法相当,或者更优的性能。此外,采用增强数据进行训练为提升药物发现过程中常见的同源系列的预测相关性和排名提供了一种高效的方法。
AEV-PLIG 还比自由能 perturbation(FEP)方法快了几个数量级,且针对每个系统的准备工作需求极少,能够生成绝对结合亲和力,而不是相对自由能差异。
这一研究突显了捕捉蛋白质-配体相互作用的新特性化潜力,以及对更强大基准的迫切需求。同时,增强数据在训练基于机器学习的评分函数方面,也在实现快速而准确的结合亲和力预测中扮演着日益重要的角色。
论文链接:https://www.nature.com/articles/s42004-025-01428-y
相关AI热点