
摘要:
最近,在生物序列建模领域,深度学习技术的发展备受关注。然而,高昂的计算需求和对大数据集的依赖给许多研究人员带来 […]
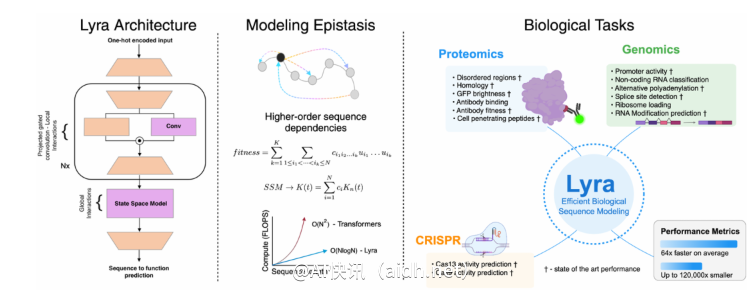
最近,在生物序列建模领域,深度学习技术的发展备受关注。然而,高昂的计算需求和对大数据集的依赖给许多研究人员带来了困扰。为解决这一难题,麻省理工学院(MIT)、哈佛大学和卡内基梅隆大学的学者联合推出了一种名为Lyra的全新生物序列建模方法。与传统模型相比,Lyra的参数数量仅为传统模型的十二万分之一,训练仅需两块GPU、仅需短短两小时,大大提高了模型的效率。
Lyra的灵感源自于生物学中的上位效应,即序列内突变之间相互作用的概念。通过次二次结构,Lyra有效地探究了生物序列与功能之间的关系。在超过100个生物任务中,Lyra展现出色的表现,包括蛋白质适应度预测、RNA功能分析和CRISPR设计等领域,有些甚至达到了目前技术的最佳水平。
与传统的卷积神经网络(CNN)和Transformer模型相比,Lyra的推理速度提高了64.18倍,同时大幅降低了参数需求。这要归功于其创新的混合模型结构,Lyra结合了状态空间模型(SSM)和投影门控卷积(PGC),以捕获生物序列中的局部和全局依赖关系。SSM通过快速傅里叶变换(FFT)高效地建模全局关系,而PGC则专注于提取局部特征,二者的结合使Lyra在计算效率和可解释性之间取得了良好平衡。
Lyra的高效性不仅推动着基础生物研究的发展,也可能在治疗开发、病原体监测和生物制造等实际应用中发挥关键作用。研究团队希望通过Lyra,更多研究人员能够在资源有限的情况下开展复杂的生物序列建模工作,从而加速生物科学的探索进程。
© 版权声明:
文章版权归作者所有,未经允许请勿转载。
本文地址:https://aidh.net/kuaixun/ut5dtbkc
暂无评论...


















